Un estudio publicado por Mireia Uribe-Herranz¹⁺², Aina Oliver-Caldés¹⁺³, Neus Martínez-Micaelo⁴, Marta Español-Rego², Maria Val-Casals¹⁺⁵, Roberto Martínez-Soler¹, Elisa Rubio-Garcia⁶⁺⁷⁺⁸⁺⁹, Valeria Brunello¹, Erik Z. Mihelic¹, Nela Klein-González¹⁺²⁺⁵, Daniel Benítez-Ribas², Núria Amigó⁴⁺¹⁰⁺¹¹, Andrea Vergara⁶⁺⁷⁺⁹⁺¹², Valentin Ortiz-Maldonado³, Luis Gerardo Rodríguez-Lobato¹⁺³, Julio Delgado³, Iñaki Ortiz de Landazuri¹⁺², Verónica González-Calle¹³, Valentín Cabañas¹⁴, Beatriz Martin-Antonio¹⁵, Lorena Pérez-Amill¹⁺⁵, Juan Luis Reguera-Ortega¹⁶, Paula Rodríguez-Otero¹⁷, Bruno Paiva¹⁷, Joaquín Martínez-López¹⁸, Maria-Victoria Mateos¹³, Mariona Pascal¹⁺², Álvaro Urbano-Ispizua¹⁺³, Europa Azucena González-Navarro², Carlos Fernández de Larrea¹⁺³, and Manel Juan¹⁺²⁺¹⁹⁺²⁰. , ha demostrado que el perfil de microbiota y metabolitos intestinales influye en la respuesta clínica de los pacientes con mieloma múltiple tratados con la terapia CAR-T dirigida al antígeno BCMA (ARI0002h).
El trabajo, publicado en Blood Cancer Discovery (2025), integra por primera vez datos de metagenómica, metabolómica e inmunofenotipado para comprender cómo el entorno microbiano intestinal condiciona la persistencia y funcionalidad de las células CAR-T.
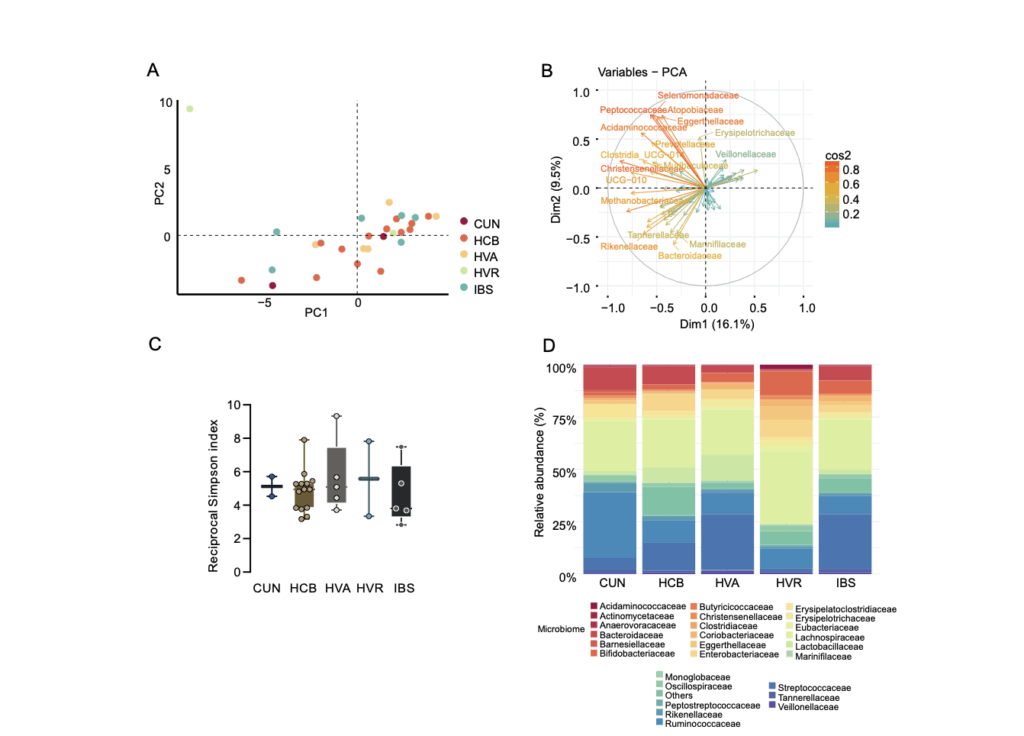
El estudio analizó muestras fecales y séricas de 51 pacientes del ensayo clínico CARTBCMA-HCB-01, recogidas en distintos momentos: antes de la aféresis, antes de la linfodepleción y tras la infusión de las células CAR-T.
Se caracterizó la composición microbiana mediante secuenciación 16S rRNA, mientras que el perfil metabolómico se evaluó por resonancia magnética nuclear (RMN) y cromatografía de gases acoplada a espectrometría de masas (GC-MS), permitiendo cuantificar un panel de 35 metabolitos fecales y ácidos grasos de cadena corta (SCFA) séricos. Paralelamente, se determinó la persistencia de células CAR-T y la respuesta clínica en distintos puntos temporales (días 28, 100 y 180).
Los autores encontraron que niveles elevados del metabolito succinato en heces antes de la infusión se correlacionaban con una mayor proporción de linfocitos CD4+ de memoria central y una mayor persistencia de las células CAR-T tras el tratamiento. En cultivos celulares, la suplementación con succinato durante la expansión ex vivo aumentó la capacidad respiratoria y el perfil metabólico asociado a mayor longevidad de las CAR-T, resultados que también se confirmaron en modelos murinos alimentados con dietas ricas en fructooligosacáridos (FOS).
Además, el análisis integrativo reveló que ciertas familias bacterianas —como Acidaminococcaceae, Barnesiellaceae y Akkermansiaceae— se asociaron a respuestas completas, mientras que Monoglobaceae y Erysipelotrichaceae se vincularon a respuestas subóptimas o ausencia de respuesta.
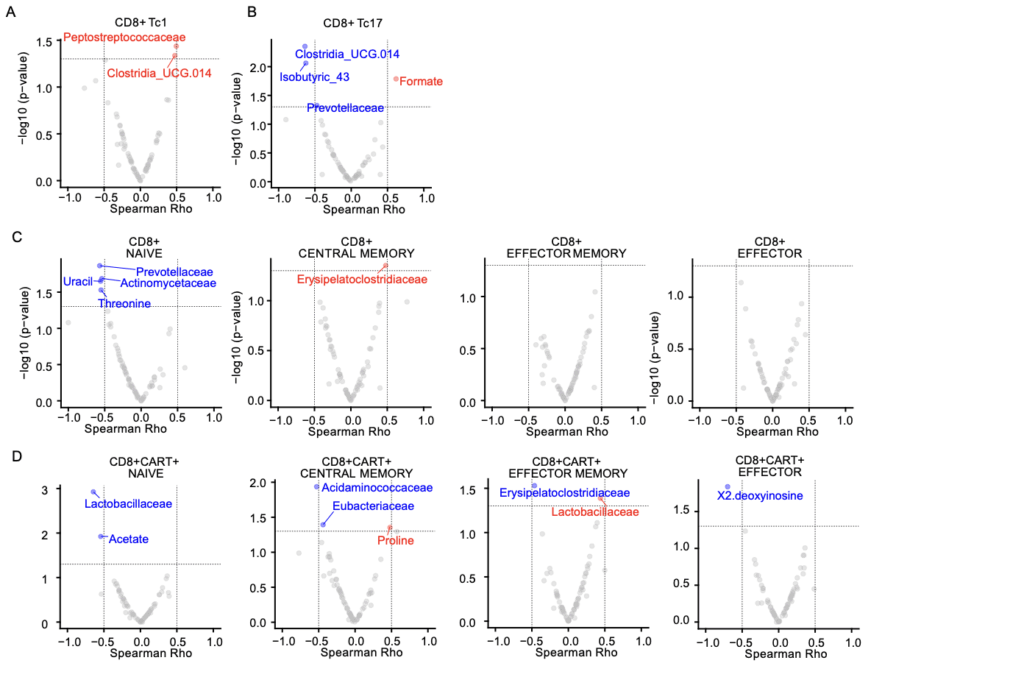
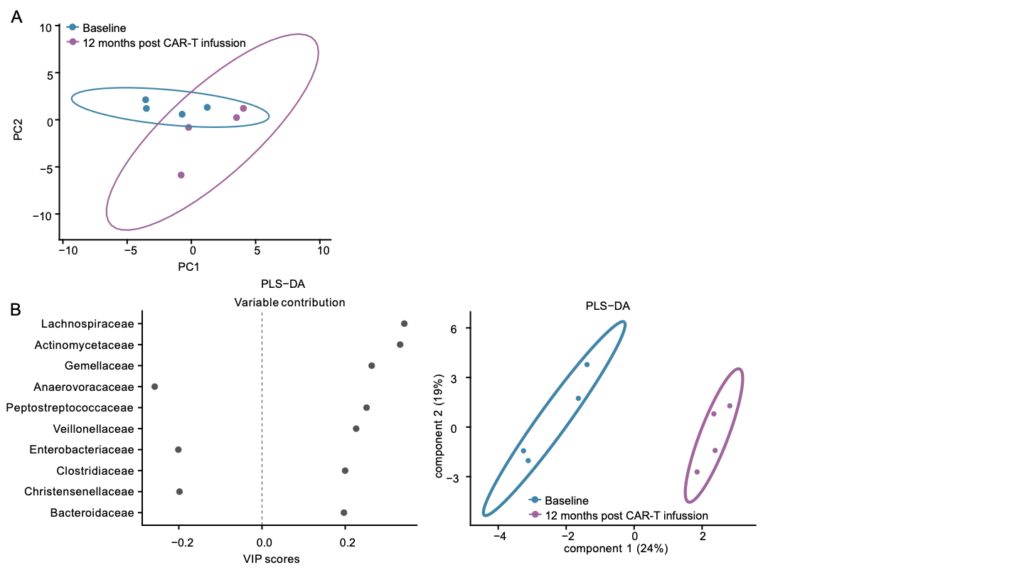
Combinando datos microbianos, metabolómicos e inmunológicos, los investigadores desarrollaron modelos multimodales de predicción de respuesta clínica, integrando información procedente de la composición bacteriana, los perfiles metabólicos y los marcadores celulares obtenidos a lo largo del tratamiento.
Mediante técnicas de aprendizaje automático y análisis multivariante, se evaluó el peso relativo de cada variable biológica en la evolución del paciente, construyendo modelos capaces de anticipar la respuesta completa con una alta precisión (AUC ≈ 0,9) tanto a los 100 como a los 180 días tras la infusión de las células CAR-T.
Estos modelos predictivos no solo permiten identificar a los pacientes con mayor probabilidad de beneficiarse de la terapia, sino que también aportan una herramienta valiosa para personalizar estrategias de tratamiento y seguimiento clínico, incorporando la influencia del microbioma y el metabolismo del huésped en la eficacia de la inmunoterapia.
En conjunto, estos resultados refuerzan la idea de que la microbiota intestinal constituye un modulador clave de la inmunoterapia celular y ponen de manifiesto el potencial del enfoque multiómico para comprender las interacciones entre metabolismo, microbioma e inmunidad antitumoral.
A futuro, el reto será trasladar este conocimiento a la práctica clínica, integrando la caracterización microbiana y metabolómica en los protocolos de selección y monitorización de pacientes tratados con CAR-T.
Asimismo, la posibilidad de intervenir sobre el microbioma mediante estrategias nutricionales, prebióticas o probióticas abre un nuevo campo de investigación orientado a potenciar la eficacia y durabilidad de las terapias CAR-T en cáncer hematológico, avanzando hacia una inmunoterapia verdaderamente personalizada y basada en la biología del paciente.
Lee el artículo completo aquí.
Institut d’Investigacions Biomèdiques August Pi i Sunyer (IDIBAPS), Barcelona, España
Departamento de Inmunología, Centre de Diagnòstic Biomèdic (CDB), Hospital Clínic de Barcelona, Barcelona, España
Departamento de Hematología, Hospital Clínic de Barcelona, Universitat de Barcelona, Barcelona, España
Biosfer Teslab, Reus, España
Gyala Therapeutics S.L., Barcelona, España
Departamento de Microbiología Clínica, CDB, Hospital Clínic de Barcelona, Barcelona, España
Barcelona Institute for Global Health (ISGlobal), Barcelona, España
Molecular Core Facility, CDB, Hospital Clínic de Barcelona, Barcelona, España
Departamento de Fonaments Clínics, Facultat de Medicina i Ciències de la Salut, Universitat de Barcelona, Barcelona, España
Departamento de Ciencias Médicas Básicas, Universitat Rovira i Virgili (URV), Institut d’Investigació Sanitària Pere Virgili (IISPV), Reus, España
Centro de Investigación Biomédica en Red de Diabetes y Enfermedades Metabólicas Asociadas (CIBERDEM), Instituto de Salud Carlos III (ISCIII), Madrid, España
CIBER de Enfermedades Infecciosas (CIBERINFEC), Madrid, España
Hospital Universitario de Salamanca, Instituto de Investigación Biomédica de Salamanca (IBSAL), Centro de Investigación del Cáncer (IBMCC-USAL, CSIC), Salamanca, España
Hospital Clínico Universitario Virgen de la Arrixaca, IMIB-Arrixaca, Universidad de Murcia, Murcia, España
Departamento de Desarrollo de Medicamentos de Terapias Avanzadas, Instituto de Salud Carlos III, Madrid, España
Hospital Universitario Virgen del Rocío, Instituto de Biomedicina de Sevilla (IBIS/CSIC/CIBERONC), Universidad de Sevilla, Sevilla, España
Clínica Universidad de Navarra, Centro de Investigación Médica Aplicada (CIMA), IDISNA, CIBERONC, Pamplona, España
Hospital Universitario 12 de Octubre, Universidad Complutense, i+12, CNIO, Madrid, España
Unidad de Inmunología Clínica, Hospital Sant Joan de Déu–Hospital Clínic de Barcelona, Barcelona, España
Universitat de Barcelona, Barcelona, España